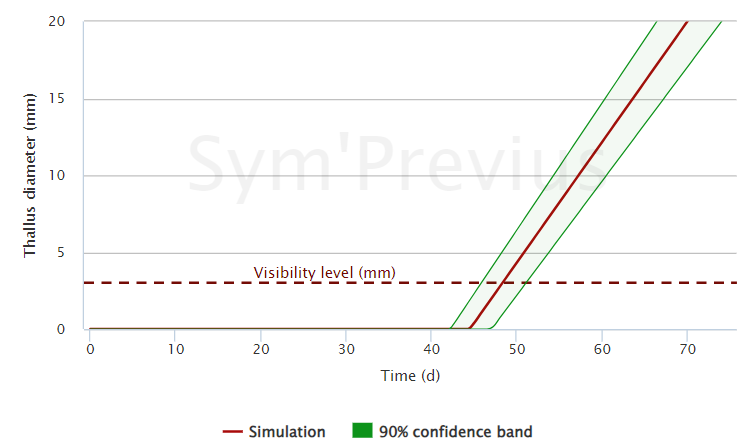
Un nouvel outil pour anticiper le développement des moisissures
Le développement des moisissures est complexe car il se produit en plusieurs étapes (germination, élongation des hyphes, prolifération mycélienne, sporulation) dont les cinétiques dépendent des facteurs environnementaux comme la température, l’activité de l’eau, la présence d’acides organiques ou de composés fongistatiques ou encore la composition de l’espace de tête dans l’emballage. Alors que la microbiologie prévisionnelle appliquée aux moisissures existe depuis de nombreuses années, aucune application visant son utilisation dans le contexte industriel n’était encore disponible. L’application en ligne Sym’Previus comporte un nouveau module fongique qui vise à combler ce manque.
L’accès à l’outil fongique est optionnel (non compris dans la version Premium standard) et requiert une souscription complémentaire.
Intéressé? Des questions ? Contactez Sym’Previus!
Un nouvel outil optimiser le couple emballage/atmosphère en fonction de l’aliment
Les interactions entre toutes les composantes sont déterminantes dans l’efficacité d’un emballage sous atmosphère modifiée. Alors, les caractéristiques des films, la composition en gaz et leur propriété de diffusion, le volume d’espace de tête, le poids et les caractéristiques de l’aliment ont été étudiés. A partir des données acquises, des modèles mathématiques ont été développés pour prendre en compte ces phénomènes, pour visualiser l’impact du choix des mélanges gazeux adaptés au produit et de l’emballage sur le développement microbiologique.
Alors que l’atmosphère modifiée est utilisée en entreprise depuis de nombreuses années, aucune application visant son utilisation dans le contexte industriel n’était encore disponible. Sym’Previus intègre désormais un nouveau module MAP (résultat de plus de 10 ans de recherche) pour faciliter la sélection des emballages adéquats et des mélanges gazeux en lien avec la formulation de l’aliment et ses conditions de stockage pour optimiser la Durée de vie des produits alimentaires.
L’accès à l’outil MAP est optionnel (non compris dans la version Premium standard) et requiert une souscription complémentaire.



